5. uzdevums
Gēnu inženierijas eksperimentu modelēšanai
paredzētā bioinformātiskas datu apstrādes rīka
"SerialCloner"
apguve.
Patstāvīgais uzdevums.
Ar bioinformātikas rīka "SerialCloner"
palīdzību izveidot gēnu inženierijas manipulāciju plānu noteikta
baktēriju gēna ievietošanai pUC grupas plazmīdu vektorā.
Mērķis - atrast piemērotākos restrikcijas enzīmus,
ar kuru palīdzību hidrolizēt kādas baktērijas genomisko DNS tā, lai
starp iegūtajiem DNS fragmentiem būtu atrodami pUC vektorā veiksmīgi
ievietojami vajadzīgo gēnu pilnā garumā (nebojātā veidā) saturoši
DNS fragmenti.
Izmantojamie vektori - pUC18 vai pUC19.
Uzdevuma izpildes gaita:
1. Iepazīties ar DNS klonēšanas vektoru pUC18 vai pUC19
uzbūvi un tajos izvietotajiem DNS klonēšanai piemērotajiem
restrikcijas saitiem (MCS rajons).
2. Iepazīties ar virtuāli klonējamā gēna DNS struktūru un
gēna tuvumā izvietotajiem DNS klonēšanai piemērotajiem restrikcijas
saitiem izvēlētajā kādas baktērijas genomiskajā sekvencē.
(atbilstošās DNS sekvences meklējamas NCBI datubāžu sadaļā "Nucleotide"), [
ja gēna nosaukumu veido vairāki vārdi, tad tos visus apvienojiet
pēdiņās: piemēram "DNA ligase
alpha" ! ]
a) izvēlieties kādu no piedāvātajām baktēriju genomiskajām vai
plazmīdu sekvencēm (pilnie genomi, genomu daļas, gēna rajonu
sekvences, plazmīdu pilnās sekvences un taml.)
[mRNS, cDNS sekvences šoreiz nebūs
piemērotas !],
b) izvēlētās sekvences apraksta daļā atrodiet
vajadzīgā gēna izvietojuma pozīcijas (nukleotīdu pozīcija no n1 līdz
n2),
c) no kopējās lielizmēra sekvences
izkopējiet ~ 12 000 bp posmu ( no pozīcijas [n1 mīnuss
aptuveni 5 000 bp] līdz [n2 + aptuveni 5 000 bp]), [ vēlams, lai izkopētajā sekvencē nebūtu
pārtraukumu vai nozīmīgi nedefinētu (NNNNN...) sekvenču posmu ! ]
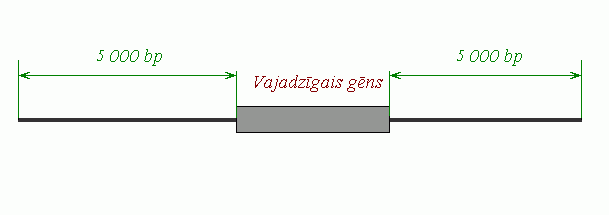
d) izkopēto sekvences daļu saglabājiet atsevišķā
failā un tālāk apstrādājiet ar bioinformātikas rīka "SerialCloner" palīdzību.
3. Paralēli veiktajām darbībām veidojiet to uzskaites un
apraksta protokolu, kura piemērs dots 1. pielikumā!
4. Izvēlēties piemērotākos restrikcijas enzīmus (-u) (A+B vai
A), ar kuriem hidrolizēt klonēšanas vektoru un klonējamo DNS, lai
izveidotu rekombinanto konstrukciju.
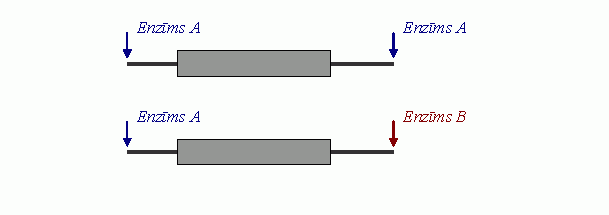
5. Izmantojot rīku "SerialCloner",
no apstrādei ņemtā genoma DNS sekvences fragmenta izdaliet ar
izvēlētajiem enzīmiem (A+B vai A) virtuāli "izšķelto" DNS sekvenci,
ko saglabājiet arī kā atsevišķu failu.
6. Ar atbilstošajiem enzīmiem virtuāli "sašķeliet" DNS
klonēšanas vektoru.
7. Sagatavoto vektoru apvienojiet ar sagatavoto insertu,
virtuāli izveidojot rekombinanto DNS molekulu, kuru saglabājiet arī
atsevišķu failu.
8. Paralēli uzdevuma izpildei veidojiet veikto darbību
protokolu!
Veikto darbību protokolam jāietver sekojoša informācija:
Izmantotais vektors.
Vektora sagatavošanai izmantotie vai izmantotais restrikcijas enzīms
un ar tiem "izšķeltā" DNS fragmenta lielums (bp) - tā DNS fragmenta
lielums, kurš netiks izmantots rekombinanta konstrukcijā, un kura
izcelsme ir no vektora (pUC18 vai pUC19).
Vektorā ievietotās DNS:
- gēna nosaukums
- saimniekorganisms (baktērijas suga, līnija)
- izmantotās genomiskās sekvences NCBI numurs un datums
- sekvences apraksts (nosaukums)
- sekvences kopējais garums (nukleotīdu pāri, bp) - izmantotās
genomiskās sekvences kopējais garums, kāds tas ir izvēlētajā NCBI
ierakstā,
neatkarīgi no tā, cik lielu šīs sekvences posmu mēs izmantosim
tālākai apstrādei
- vajadzīgā gēna izvietojums (nukleotīdu pozīcija izvēlētajā NCBI
ieraksta sekvencē, kas parāda vajadzīgā gēna sākumu un beigas: no -
līdz)
- vajadzīgā gēna garumu (bp: = gēna beigu pozīcija mīnus gēna sākuma
pozīcija+1)
- vajadzīgā gēna kodētā proteīna garums
(aminoskābju atlikumu skaits proteīnā)
- izmantotais sekvences posms (nukleotīdu pozīcija izvēlētajā NCBI
ieraksta sekvencē no - līdz) [no pozīcijas [n1-5 000] līdz [n2+5
000] - tālākajai apstrādei
izraudzītā (izkopētā) DNS posma pozīcijas attiecīgajā NCBI ierakstā
esošajā pilnā garuma sekvencē
- vajadzīgā gēna izvietojums izmantotajā sekvences posmā (parāda
vajadzīgā gēna sākuma un beigu pazīciju: no - līdz sekvences posmā,
kurš tiks izmantots apstrādei "SerialCloner")
- vektorā insertējamās sekvences sagatavošanai izvēlētie un
izmantotie restrikcijas enzīmi
- ar šiem enzīmiem no izmantotā sekvences posma "izšķeltās"
sekvences garums (nukleotīdu pāri, bp).
No NCBI sekvences tālākai apstrādei izvēlēto (izkopēto) sekvences
daļu saturošā faila nosaukums.
No izvēlētās (izkopētās) sekvences virtuāli izšķeltās sekvences
faila nosaukums
Ja veiktas kādas vektora vai insertējamās DNS galu modifikācijas,
noteikti norādiet arī tās!
Virtuāli iegūtā DNS rekombinanta (sagatavotais vektors +
sagatavotais inserts) DNS kopējais izmērs (nukleotīdu pāri, bp)
Virtuāli radītā rekombinanta DNS sekvences faila nosaukums.
Izpildīts patstāvīgais darbs ietver:
1. Veikto darbību protokolu;
2.
Aatsevišķu failu, kurš satur no NCBI sekvences tālākai apstrādei
izvēlētā (izkopētā) posma sekvenci pilnā garumā.
3. Aatsevišķu failu, kurš satur no izvēlētās (izkopētās) sekvences
virtuāli "izšķeltā" DNS fragmenta sekvenci (tai skaitā ietver arī
virtuāli klonējamā gēna sekvenci).
4. Aatsevišķu failu - virtuāli izveidotās rekombinantās DNS
(sagatavotais vektors + sagatavotais inserts) sekvenci.
Kad gatavs, viss iepriekš uzskaitītais sapakojams
arhīvā (!!! [citu gadu
bēdīgā pieredze rāda, ka e-pasti ar nearhivētām DNS
sekvencēm mēdz pazust]) un
sūtams uz adresi:
1. pielikums
Veikto darbību protokols (aizpildīts
piemērs).
Izmantotais vektors: pUC19
vektora sagatavošanai izmantotie restrikcijas
enzīmi:
HindIII
Ecl136II
Tie no vektora
"izšķeļ"
45 bp lielu DNS fragmentu
Vektorā ievietotā DNS:
gēna
nosaukums
invertāze
saimniekorganisms (baktērijas suga,
līnija) E. coli ATCC 1483
NCBI sekvences nummurs un
datums
NT_ADD010050007
13-NOV-2012
sekvences apraksts
" E. coli genome shotgun sequence 007"
sekvences kopējais
garums
837 287 bp
nepieciešamā gēna pozīcijas šajā
sekvencē
no 154 076 līdz 155 351
gēna
garums
1 275 bp
gēna kodētā proteīna
garums
425 aminoskābes
izmantotais sekvences posms (nukleotīdi no -
līdz) 149 061 - 160 350
nepieciešamā gēna pozīcijas izmantotajā sekvences posmā (nukleotīdi
no - līdz)
no 5 015 līdz 6 290
inserta sagatavošanai izmantotie restrikcijas
enzīmi HindIII
PvuII
"izšķeltā" inserta sekvences
garums
2 683 bp
DNS galu modifikācijas:
vektoram
nav veiktas
insertam
nav
veiktas
virtuāli iegūtās rekombinantās DNS
izmērs 5 324 bp
Pievienoto
sekvenču failu nosaukumi:
Izmantotā (No NCBI izkopētā) DNS posma
sekvence
InvRajons.txt
No izmantotās (izkopētās) sekvences ar izvēlētajiem enzīmiem
virtuāli "izšķeltā" DNS fragmenta sekvences faila nosaukums
InvFr13.xdna
Virtuāli iegūtās rekombinantās DNS faila
nosaukums
pUC19Inv13.xdna

